-
文章 · 2026年02月08日
选择 Opentrons Flex 的 7 大理由
阅读全文 -
文章 · 2026年02月08日
Opentrons Flex 实用操作技巧:新手必读
阅读全文 -
文章 · 2026年02月07日
高通量 NGS 建库流程中的关键步骤与 Opentrons Flex 支持
阅读全文


大规模 SARS-CoV-2 基因组测序对于追踪当前疫情期间的病毒进化至关重要。最早在英国、南非和巴西发现的变异株已传播到多个国家。我们开发了软件工具“变异数据库 (VDB)”,用于快速检查刺突突变的变化情况。使用 VDB,我们在纽约地区发现了一种新兴的 SARS-CoV-2 谱系,其与之前报告的变异株有相同的突变。该谱系(现指定为 B.1.526)中最常见的刺突突变组是 L5F、T95I、D253G、E484K 或 S477N、D614G 和 A701V。该谱系于 2020 年 11 月下旬首次测序,当时它占纽约市 (NYC) 收集的已测序冠状病毒基因组的不到 1%。到 2021 年 2 月,该谱系的基因组占纽约市标本 3288 个测序基因组的约32 %。系统发育动力学推断证实了 B.1.526 谱系在纽约市的快速增长,尤其是由刺突突变 E484K 定义的亚支系,其增长速度超过了纽约市其他变体的增长速度。假病毒中和实验表明,B.1.526 刺突突变对恢复期和疫苗接种者血浆的中和滴度产生不利影响,表明该谱系对公共卫生的重要性。
介绍
在 2020 年 SARS-CoV-2 大流行的最初几个月之后,绝大多数测序基因组都含有刺突突变 D614G(以及 3 个独立的核苷酸变化)1。经过一段时间的逐渐变化后,2020 年第四季度出现了几种包含多个突变的变体,其中许多位于刺突基因内2 – 5。多种证据表明,逃避抗体选择压力是这些变体发展的驱动力6 – 9。
目前,SARS-CoV-2 的基因组监测重点是监测这些变体的出现以及它们的突变可能对被动抗体疗法的有效性和疫苗预防轻度或中度 COVID-19 的有效性产生的功能性影响。虽然越来越多的样本正在被测序,但对这些基因组的分析仍然是一个挑战10。在这里,我们开发了一个简单快捷的实用程序,可以快速检查 SARS-CoV-2 基因组监测揭示的突变景观:变体数据库 ( vdb )。利用此工具,我们发现了几组最近测序的基因组,这些基因组在关键抗体表位上发生了突变。在这组中有一个在纽约市出现的新谱系,其频率有所增加,截至 2021 年 2 月,已占测序基因组的约32%。我们通过系统发育推断证实了 B.1.526 在 2021 年初在纽约市的迅速传播。此外,我们评估了 B.1.526 刺突突变对恢复期和疫苗接种者血浆中和滴度的影响。
结果
系统发育分析对于了解病毒基因组的关系至关重要。然而,其他视角也有助于在大量序列中检测模式。我们开发了vdb作为一种实用程序来查询基因组监测期间观察到的尖峰突变集。使用vdb工具分析全球禽流感数据共享倡议 (GISAID) 数据集中的 SARS-CoV-2 序列11,12 ,我们检测到了几个不同于变体 B.1.1.7、B.1.351、B.1.1.248 和 B.1.429 2 – 5的序列簇,这些序列簇在已知与抗 SARS-CoV-2 抗体相关的位点上发生尖峰突变8,13 (表1)。vdb程序可以找到具有相同尖峰突变集的病毒簇,然后可以使用这些模式来查找潜在相关的序列。
定义 B.1.526 的突变
从纽约地区收集了一个值得注意的基因组序列簇,代表了一个独特的谱系,现被指定为 B.1.526(图 1,补充图 1)。该变体存在于 20.C 进化枝中,以 3 个定义的尖峰突变为特征:L5F、T95I 和 D253G。在 B.1.526 中,最大的亚进化枝由 E484K 定义,两个不同的亚进化枝分别由 S477N 定义;这两个突变都位于尖峰的受体结合域 (RBD) 内(图 2和补充表 1)。我们注意到,尖峰位置 701 的进化历史取决于该树是使用分子钟生根(图 1)还是其姐妹进化枝(以 L452R 突变为特征;补充图 2),后者假设替换 A701V,然后是回复 V701A。在 B.1.526 谱系中的核苷酸突变中,最具特征性的突变包括 A16500C (NSP13 Q88H)、A22320G (spike D253G) 和 T9867C (NSP4_L438P)。B.1.526 谱系的另一个显著特征是核苷酸 11288-11296 (NSP6 106-108) 的缺失,这种现象也出现在变体 B.1.1.7、B.1.351、P.1 和 B.1.525 14中。
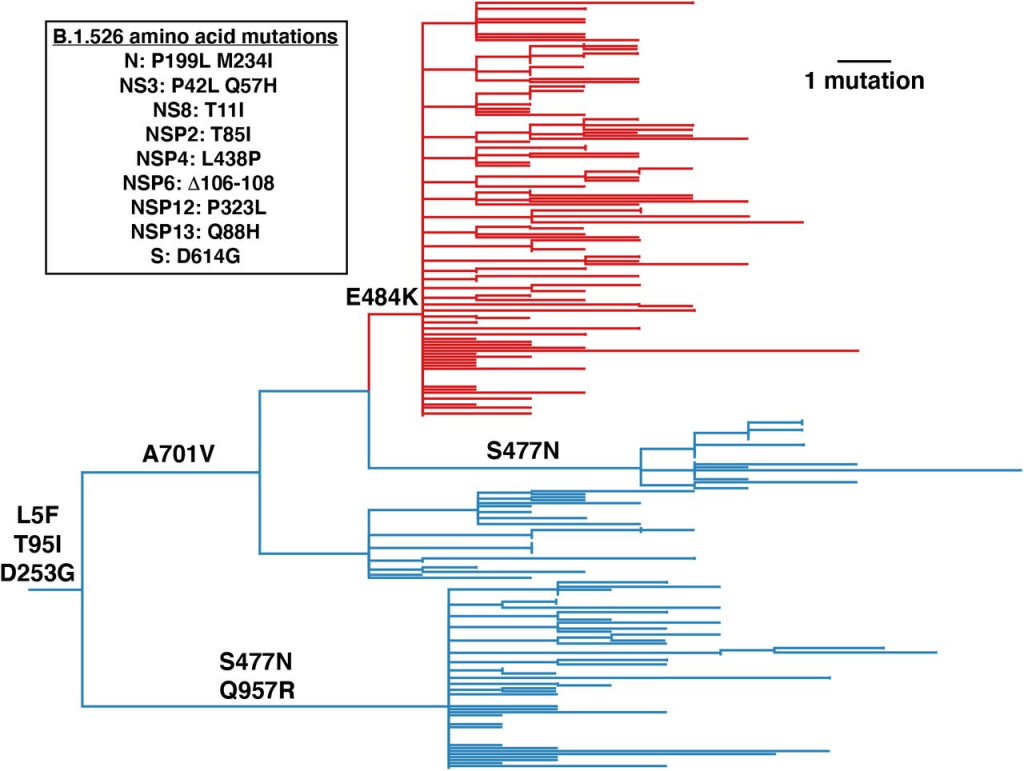
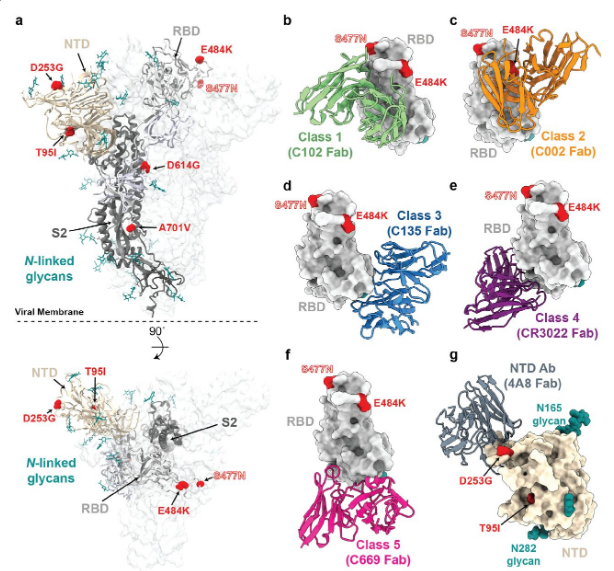
关于该谱系中流行的四种刺突突变:(1) 已知 E484K 可减弱多种抗 SARS-CoV-2 抗体的中和作用,特别是在 2 类抗 RBD 中和抗体中发现的抗体13、15 ,并且也存在于变体 B.1.351 4和 P.1/B.1.1.248 2中,(2) 据报道,D253G 是针对 N 端结构域抗体的逃逸突变16,(3) S477N 已在几个早期谱系中被鉴定17,靠近多种抗体的表位18,并且已被证明通过增强与 ACE2 的相互作用来增加病毒的传染性19、20,以及 (4) A701V 位于相邻原体的 S2' 裂解位点附近,并与变体 B.1.351 4共享。 B.1.526 谱系的总体突变模式(图 2)表明,该谱系的出现部分是由于抗体的选择压力。根据这些病毒的收集日期,似乎该谱系在纽约的频率迅速增加。
经验丰富的服务团队和强大的生产支持团队为客户提供无忧的订单服务。

简体中文

繁體中文

English

日本語

한국인